化学学院教授罗琼以最后通讯作者在《美国国家科学院院刊》(PNAS)发表了题为《Linking DNA-packing density distribution and TAD boundary locations》的研究论文。该论文首次揭示染色质物理特性中的DNA压缩密度是驱动染色质拓扑关联结构域(Topologically Associating Domains,TADs)形成的关键因素,为理解基因组三维结构形成机理提供了全新视角。该研究由华南农业大学孟露明、香港科技大学常富杰和华南师范大学化学学院罗琼共同完成,华南师范大学美术学院副教授陈瀚对论文的作图提供了艺术指导。
基因组三维结构为基因表达提供了物理平台,其动态调整确保了细胞在不同发育阶段和功能状态下能够精准执行生物学任务。研究发现,在真核细胞的间期细胞核中,基因组三维结构的单元是拓扑关联结构域(Topologically Associating Domains,TADs )。TADs一般覆盖千到百万碱基的染色质区域,其内部具有较强的相互作用,而边界区域则常富集有cohesin和CTCF等蛋白。TAD边界具有绝缘作用,能够阻隔不同TAD之间的相互作用,即能有效阻断异常的增强子-启动子接触,从而维持正常的基因表达调控。因此,TAD形成机制一直是生命科学关注的核心问题之一。当前,学界普遍认为由cohesin介导的环挤压(Loop Extrusion,LE)过程是TAD形成的主要机制。众多发表在顶级期刊如Nature、Science和Cell等的研究都致力于观察LE模型描述的生物化学过程。遗憾的是,该过程尚未在体内直接观察到。这导致LE模型的关键参数(如cohesin的挤压速度和停留时间)需要人为设定,并在不同细胞类型中不同,这使得预测基因组三维结构动态变化的问题具有挑战性。
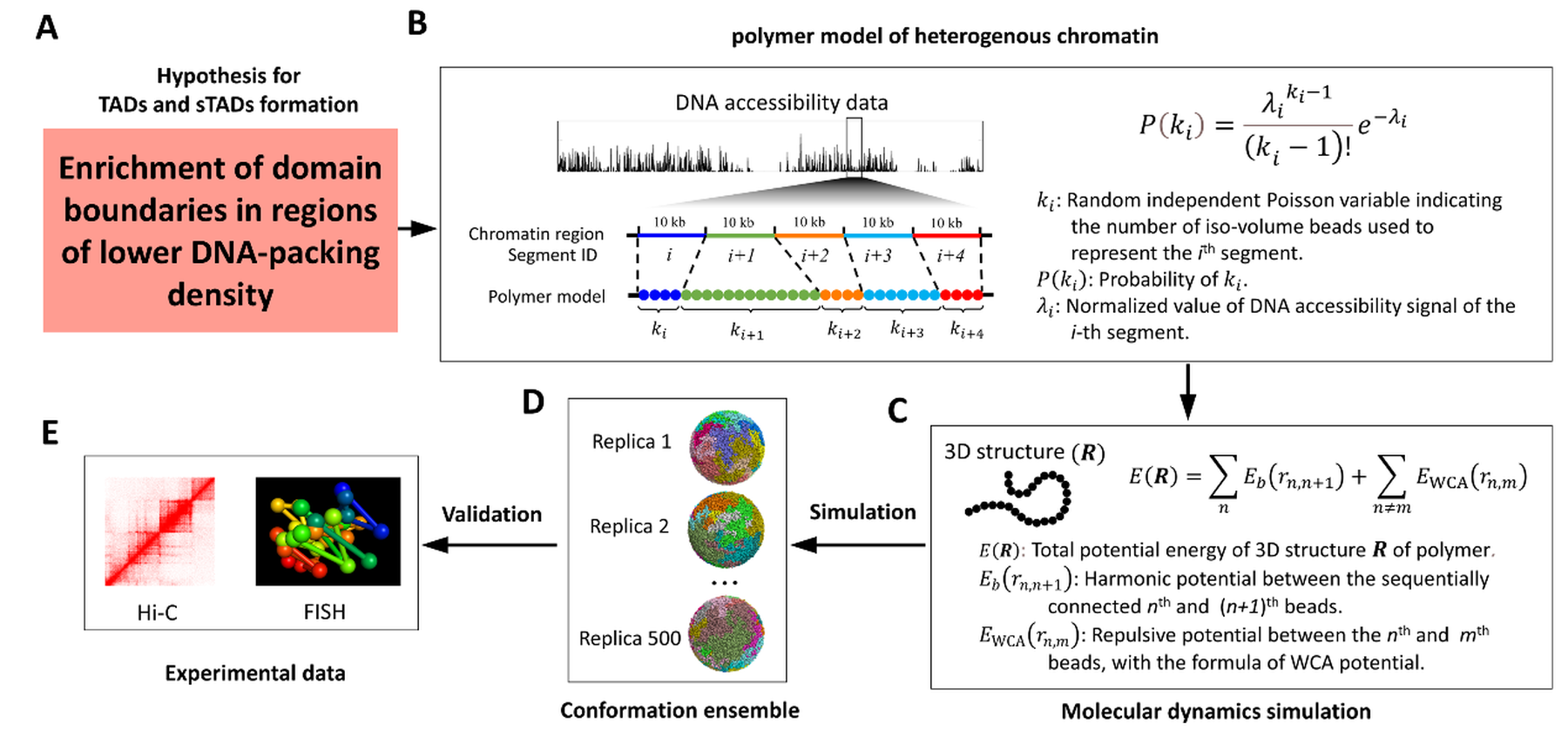
罗琼等提出“染色质的物理压缩状态主导了TAD边界的形成”这一新观点。为了验证这一假设,他们开发了使用ATAC-seq和DNase-seq等染色质可及性数据参数化DNA压缩密度的新算法,自编了能在全基因尺度上模拟染色质折叠的分子动力学软件,得到了首个 “DNA压缩密度——三维结构”预测模型。该模型特点为:(1) 无需预设参数:仅利用实验可测的染色质可及性数据量化DNA压缩密度;(2) 高预测精度:成功重构人类基因组中60%以上的TAD边界;(3)动态追踪能力:首次预测了T细胞早期分化中关键TAD边界的实时变化。
这项工作建立了染色质物理特性与染色质三维结构之间的联系,为理解染色质三维结构形成机制提供了全新的视角。同时,也为利用染色质可及性数据预测全基因组TAD结构提供了可行的技术路径和方法,有望突破现有研究中关于预测染色质三维结构动态变化的技术瓶颈。该模型将可能成为解析染色质三维结构与基因调控之间内在联系,以及探索细胞分化和病理状态下基因组三维结构变化的重要工具,为生命科学和医学健康领域的相关研究提供更加精准的理论和方法支持。
该模型获得授权发明专利:ZL 2022 1 0736148.6
论文链接:https://www.pnas.org/doi/10.1073/pnas.2418456122